|

|

|

|
|

|
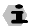 > Bilderklärung - DNA-Sequenz von Arabidopsis <
> Bilderklärung - DNA-Sequenz von Arabidopsis <
|
Ausschnitt einer codierenden DNA-Sequenz von Arabidopsis
Ein Stück DNA eines Gens aus der Forschungspflanze Arabidopsis thaliana (Ackerschmalwand) wurde zur Bestimmung der Basenreihenfolge mit Hilfe der Kettenabbruchmethode nach Sanger automatisch sequenziert. Das Bild zeigt das Resultat der Analyse, wobei die nummerierte Reihenfolge der Nukleotide (A=Adenosin, T=Thymidin, C=Cytosin, G=Guanosin) dargestellt ist.
Kettenabbruchmethode nach Sanger
Bei der Zellteilung müssen für die Tochterzellen jeweils identische DNA-Kopien angefertigt werden. Dabei dienen die DNA-Einzelstränge als Vorlage, um einzelne Nukleotidbausteine zu neuen Strängen zusammenzufügen. Das Prinzip von Sanger beruht darauf, dass bei diesem Kopierprozess nicht nur gewöhnliche Nukleotide bereitgestellt werden, sondern auch chemisch veränderte, bei denen der Kopiervorgang abbricht. Diese Nukleotide tragen gleichzeitig eine Fluoreszenzmarkierung, und zwar eine von vier Farben je nach der Art ihrer Base.
Wenn ein DNA-Strang nach dem Prinzip von Sanger kopiert wird, dann entstehen also DNA-Ketten unterschiedlicher Länge, die jeweils an ihren Enden farbig markiert sind. Diese DNA-Ketten werden mit Hilfe der Gelchromatographie nach Grösse getrennt. Die DNA-Sequenz des Stranges kann dann direkt bestimmt werden, indem ein Laser-Scanner über das Gel fährt und die Reihenfolge aller endständigen und farbig markierten Nukleotide erfasst. Das Sequenzieren von DNA ist heute automatisiert und leistungsfähige Geräte können pro Stunde mehrere tausend Basen bestimmen.
|
|
|
|



